翻译组反映了mRNA翻译合成蛋白质的过程,包含基因表达过程在翻译层级的重要信息。核糖体分析(ribosome profiling)是翻译组研究的主流技术,通过对核糖体保护片段(RPF)的深度测序,对翻译效率进行定量,在亚密码子精度下解析翻译的实时状态。
近年来非常活跃的翻译研究领域积累了大量核糖体分析数据,描绘了各种条件下(如药物处理、基因敲除或敲低、各种压力环境等)细胞翻译组的异常改变。此前的研究通常只针对特定实验条件下的翻译组,而杨雪瑞课题组则提出,大量不同的实验条件下产生的数据通过整合形成大数据集,提供了翻译组复杂变化的多样性,为翻译调控模式的研究提供了高信息量的参考坐标框架。
使用这一策略,杨雪瑞课题组首次揭示化合物RocA通过翻译起始因子eIF4A调控翻译延伸过程的新功能。这一成果于2023年2月2日发表于Nature Communications,文章题目:核糖体分析大数据集的重新分析揭示RocA通过eIF4A蛋白对翻译延伸动态的干扰作用(Reanalysis of ribosome profiling datasets reveals a function of rocaglamide A in perturbing the dynamics of translation elongation via eIF4A)。
RocA是楝科植物中发现的天然化合物,具有非常好的抗肿瘤、抗病毒功能。RocA及其衍生物是近年来临床研究的热门分子。此前的研究证明RocA通过其靶点蛋白eIF4A,阻碍43S翻译前起始复合物PIC在5’ UTR上的扫描,从而抑制翻译起始。在杨雪瑞课题组的研究中,他们首先收集整理了100余套人类细胞在不同药物或基因扰动条件下的核糖体分析数据,并使用此前开发的翻译组数据解析工具,定量分析了细胞翻译起始与延伸动态过程的变化,形成覆盖各种翻译调控模式的参考坐标系。其中一个非常令人意外的观察是,作为众所周知的翻译起始抑制剂,RocA却与其它经典的翻译延伸调控作用聚为一类。
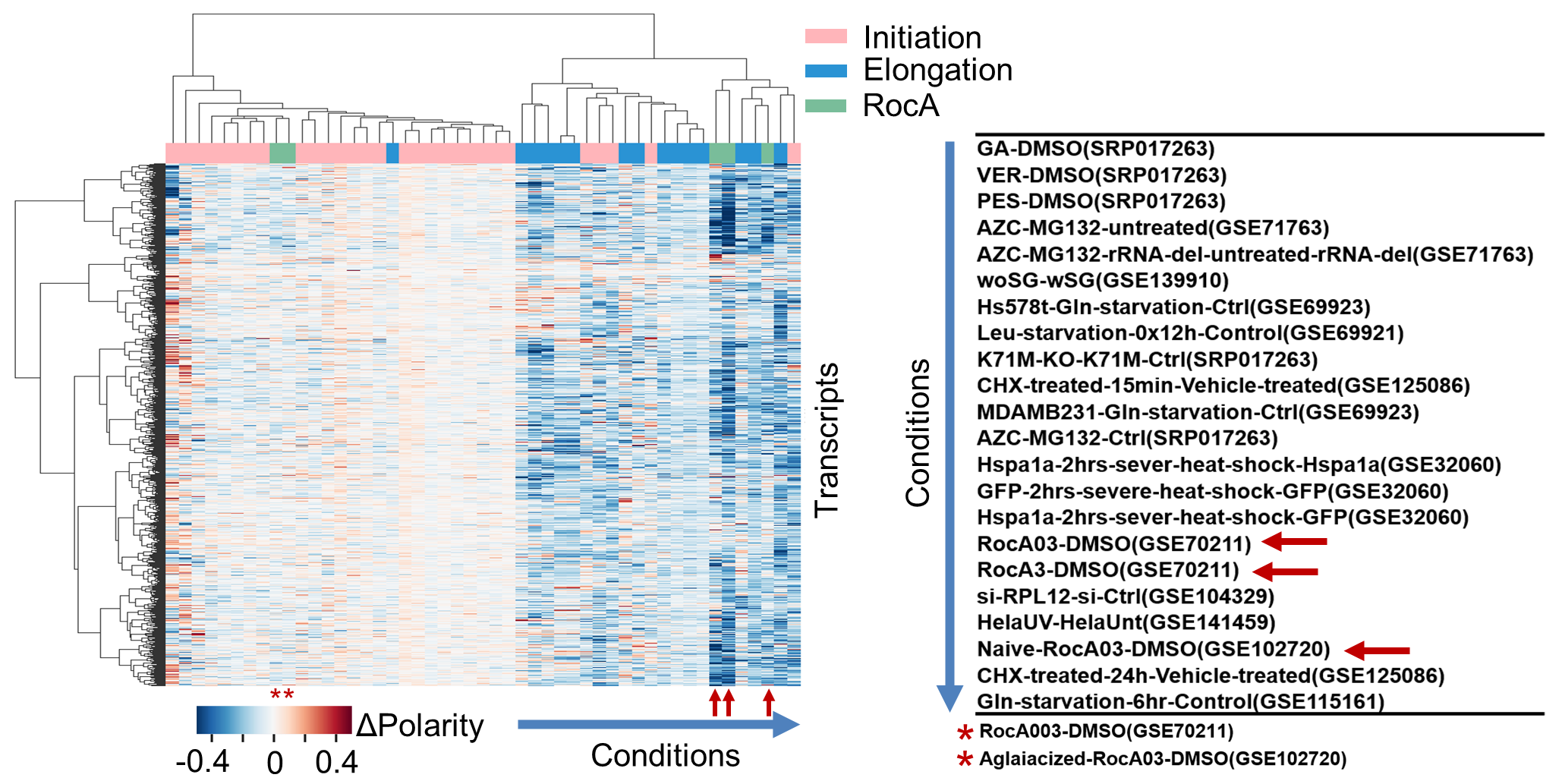
图1:翻译组大数据的整合分析揭示不同的翻译调控模式
在这一关键信息与线索的启发下,通过对一系列核糖体分析数据的深入挖掘,研究最终揭示了RocA双模态、选择性的翻译抑制功能,即一部分基因转录本的翻译被抑制于翻译起始阶段,而另一部分抑制于翻译早期延伸阶段。通过对eIF4A突变的核糖体分析数据及eIF4A iCLIP数据的分析,研究证明两个作用机制都依赖于eIF4A与RNA 5’ UTR及CDS区多嘌呤序列的结合。进一步,disome-seq分析证明RocA通过eIF4A引起核糖体在翻译早期延伸阶段的累积,引起核糖体碰撞,阻滞翻译延伸。
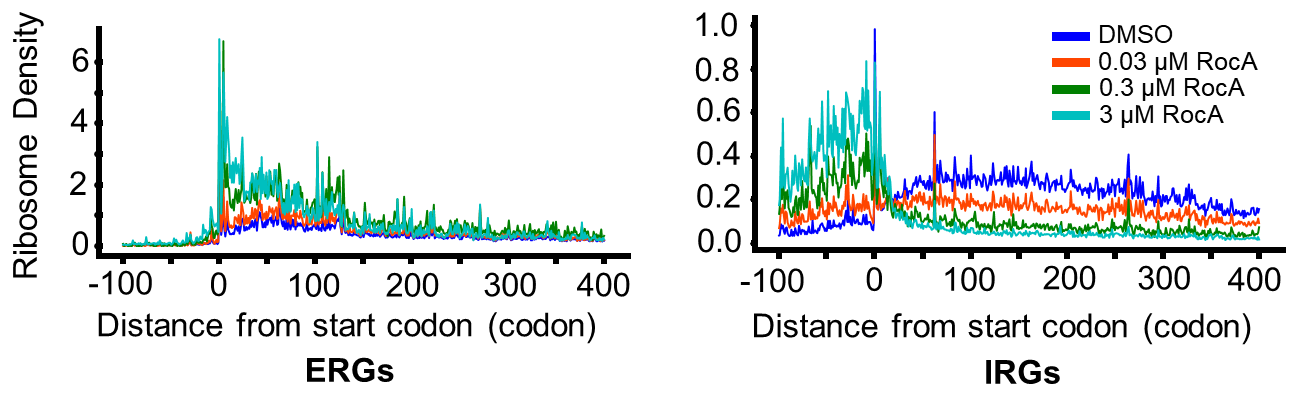
图2:RocA对两种基因的双模态翻译调控模式(定义为延伸响应型基因ERGs与起始响应型基因IRGs)
本研究发现RocA引起的核糖体累积与翻译延伸阻滞在多种肿瘤细胞中都存在,因此进一步提出RocA抑制肿瘤的新机制,即RocA通过eIF4A抑制翻译延伸,通过阻滞核糖体移动,导致disome形成。而此前的研究已经证明异常累积的核糖体碰撞形成的disome会招募蛋白激酶ZAKα,通过激活压力感知激酶p38和JNK来诱导细胞的凋亡。
杨雪瑞课题组长期关注复杂生物学过程中的翻译组及其调控,开发了一系列针对核糖体分析数据的算法工具,从不同角度深入解析翻译组图谱与翻译动态调控过程(Nat Commn, 2016; NAR, 2018; BMC Bioinfo, 2020)。这些工具支持了一系列合作研究,帮助解析了多个重要生物学过程中的特异性翻译调控机制(Cell Res, 2017, 2021; Mol Cell, 2020)。此次发表的论文进一步展示了一种有特色的研究模式,即高效的数据挖掘算法与大数据资源结合,产生新线索,进一步在分子机制研究的支持下,获得新的生物学发现。
清华大学生命学院2017级博士生李发金、2016级博士生方剑火、2020级博士生于奕凡为论文共同第一作者,杨雪瑞副教授与李发金为论文通讯作者。研究工作得到国家自然科学基金委、国家重点研发计划重点专项的资助。国家蛋白质科学研究(北京)设施(清华大学蛋白质研究技术中心)下属生物计算平台、基因测序与分析平台为本研究提供了大力支持。


